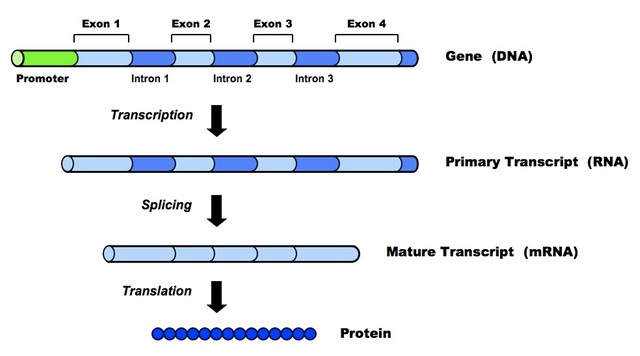
마구잡이 정리
- Genomic interval – Tuple (chromosome, strand, start poisition, end position)
- Genomic region – Ordered set of genomic intervals
- 유전체 (genome) – Gene + Chromosome; 특정 개체의 염색체에 포함된 모든 유전물질(DNA)을 통틀어 부름
- 유전체 지도 – genomic mapping
- 유전마커
- microsatellite (STR; short tandem repeat)
- http://ib.bioninja.com.au/_Media/str_med.jpeg
- SNP; single nucleotide polymorphism
- 전체 인간 유전변이형의 90%를 담당
- 인간 집단에서 1% 이상의 빈도로 존재
- 1% 이상 되지 않는 경우 mutation이라고 부름
- 최근에는 빈도에 상관없이 SNV(variant) 라고 부름
- RFLP: restriction fragment length polymorphism – 대립유전자형 마커
- microsatellite (STR; short tandem repeat)
- 유전지도 1 cM(centiMorgan) : 두 마커간에 1%의 재조합이 발생하는 거리
- Linkage mapping : 유전마커 M1,M2 를 통해 분석한 유전병이 재조합율을 통해 n% 일 경우로 나타날 경우 이떄 가설에 합당한 유전마커(예:M1)로 부터 n cM 떨어져있다고 볼 수 있다. (유전자 찾는 방법)
- 동일한 물리적 거리에 유전자 재조합이 많이 일어나는 부위가 포함되어있다면 그 거리보다 유전적 거리가 더 크게 표시될 수 있다.
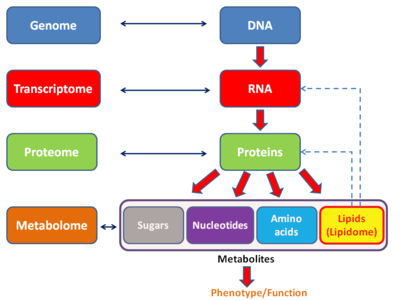
- Microarray analysis – Pathway 분석은 DAVID(https://david.ncifcrf.gov/) 또는 Ingenuity Pathway Analysis 등을 이용한다.
- NGS와 DNA microarray
- DNA microarray는 특정 유전자에 대한 발현의 정도만 분석(정량적)
- NGS – 유전변이, 유전자 발현량, 대립유전자형에 대한 상대적 발현차이(allelic gene expression), alternative splicing 변이형, fusion gene을 포함한 새로운 전사체 발굴 등에 활용
- STR 마커 vs SNP 마커
- STR과 같은 microsatellite 마커는 다양한 대립 유전자(multi-allelic)이 존재 > 다양한 대립 유전자란 ABO 혈액형 처럼 2개 이상의 대립유전자가 결합한다는 의미
- 흔히는 2개 염기서열 반복이 흔하다(CACACA등)
- 세대를 거쳐 유전 > 개인 식별에 활용, 유전자 감식, 친자 확인 등
- SNP는 두 종류의 대립인자(bi-allelic)으로 존재함
-
- 두 대립 유전자(CCATG – CCACG) 이렇게 있을 경우 두 종류 중에 더 빈도가 낮은 것을 기준으로 분류한다. (즉 MAF; minor allele frequency의 최대값은 0.5가 된다)
- rSNP, iSNP, gSNP, cSNP, sSNP, nsSNP
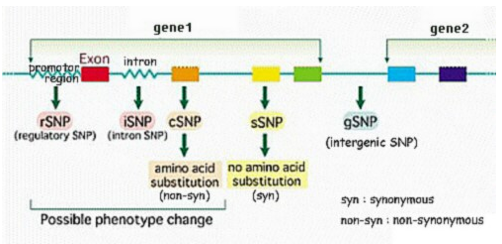
출처: https://dic1224.blog.me/80186095417 - nsSNP의 경우 다른 SNP에 비해 적게 존재, 빈도 낮음 > 아미노산 변화를 초래하는 경우 자연탈락된다는 가설(자연 선별)
-
- STR과 같은 microsatellite 마커는 다양한 대립 유전자(multi-allelic)이 존재 > 다양한 대립 유전자란 ABO 혈액형 처럼 2개 이상의 대립유전자가 결합한다는 의미